每月初,创腾科技生命科学模拟技术部都会通过微信公众号定期总结 BIOVIA Discovery Studio 用户在软件使用过程中产生的一些具有代表性的问题。基于我们的工程师团队在分子模拟领域内十多年的经验,定期为大家带来实实在在的技术干货分享。
本月的技术干货Q&A分享我们将给大家带来在同源建模中常遇到的问题:
1. 进行同源建模时能否使用自己挑选和下载的多模板进行建模?如果可以,应如何操作。
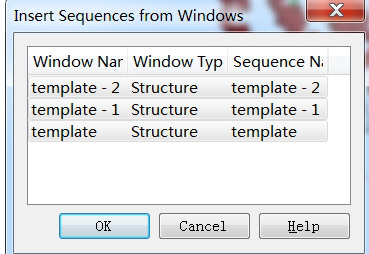
答:使用自己挑选的模板可以直接将BLAST这步跳过,可以直接打开自己挑选的多个模板,然后在目标序列窗口任意位置,鼠标右键点击Insert sequence,然后选择from windows,选中模板,点击ok即可。然后再将进行序列比对就可以了,剩余其他步骤都是一样的。
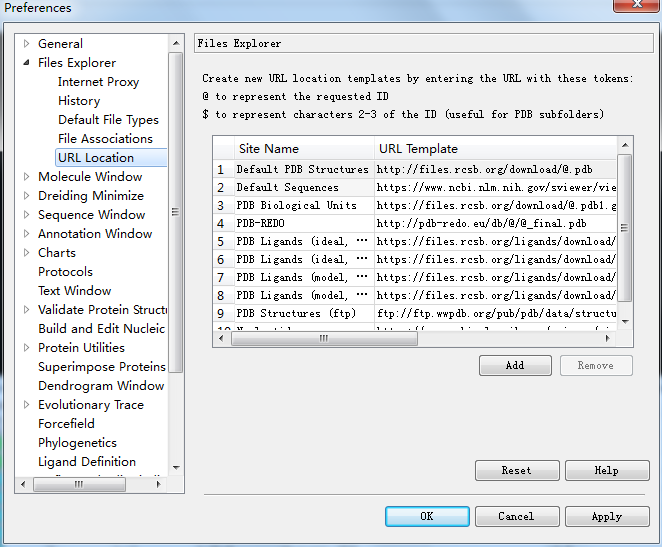
2. 同源建模无法进行Load Structure and Alignment
答:可以通过菜单Edit — Preference更改一下PDB下载链接,把https改为http试试,或者http改为https。
3. 同源建模结束后,拉式图评估结果显示有氨基酸需要优化,应该怎么优化呢?
答:可以先通过结构查看不合理氨基酸是否在关键位置,如果不在可以不用优化,如果在关键位置,则可以根据氨基酸所在位置采用Loop Refinement、Side-Chain Refinement、Minimization或者分子动力学模拟优化也是可以的。
4. 同源模建时出现下述错误,该如何解决?
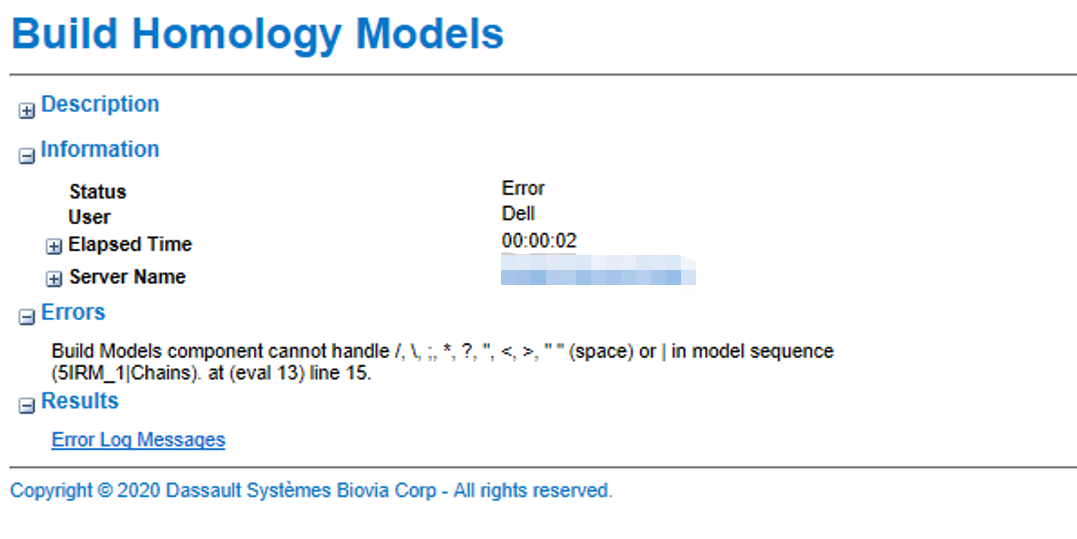
答:错误显示建模时使用的序列或输入文件中有程序不识别的字符,BIOVIA Discovery Studio不能识别中文以及一些特殊符号,建议删除相关字符后再进行建模。
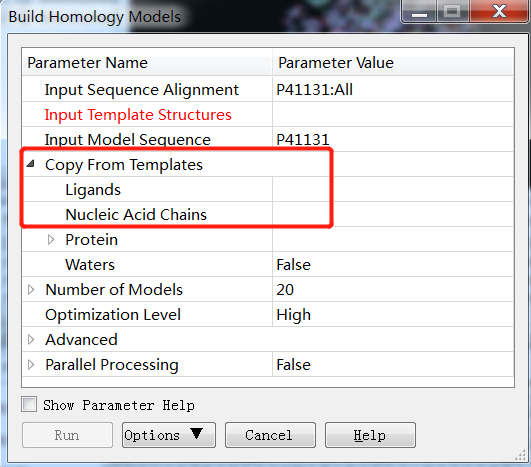
5. 想建模后的结构中带有辅因子,如葡萄糖氧化酶GOD带有黄素腺嘌呤二核苷酸FAD,若基于一个已知的晶体结构(带有FAD的GOD酶),如何得到带有FAD的自有序列的蛋白模型呢?
答:建模时选择带有FAD的蛋白模板,建模参数设置Copy From Sequence即可。