
Discovery Studio 2020 相较于Discovery Studio 2019进行了一系列产品性能的提高和新功能的增加,这些功能进一步扩展了研究的热点领域,例如添加显式膜结构的跨膜蛋白分子动力学研究和抗体成药性评价等。
新功能涉及分子动力学(Simulation)、药效团模型(Pharmacophores)、蛋白处理(Macromolecules)、抗体设计(Antibody)、分子对接(Receptor-Ligand Interactions)和毒性预测(TOPKAT)并进行更新和改进。

分子动力学(Simulation):
l 新增Multi-Site Lambda Dynamics method(MSLD):用于计算大型类属库分子的相对结合自由能,更好地进行先导化合物的筛选优化;
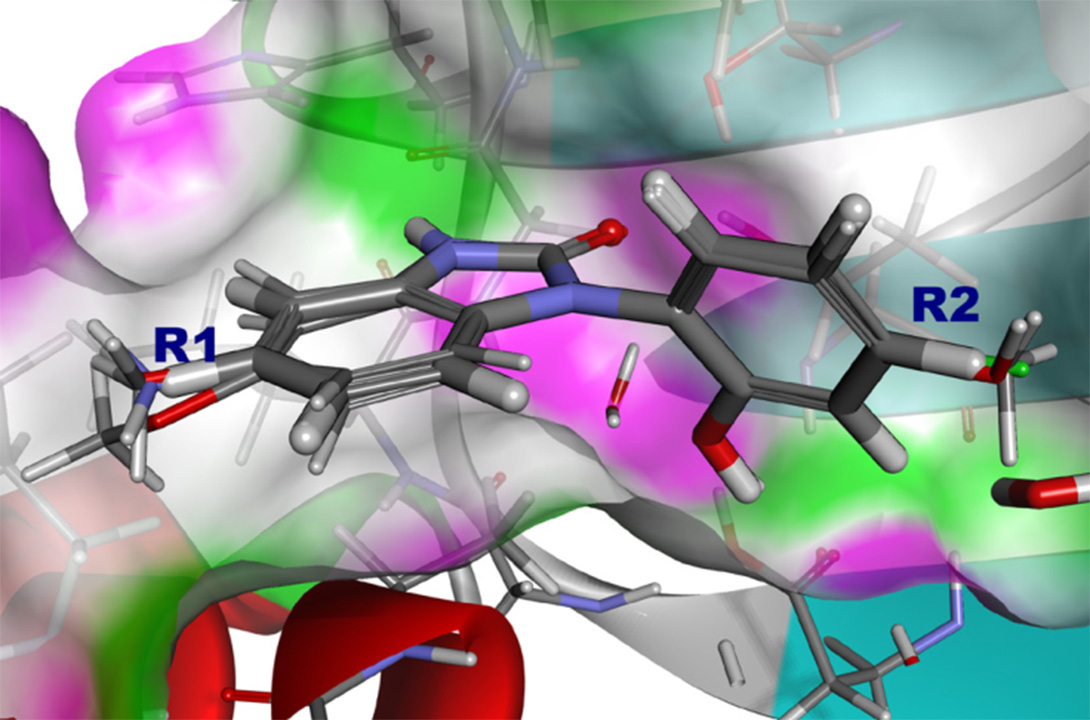
图1:通过MSLD方法计算组合库中分子的相对结合自由能
l 新增显式膜结构的跨膜蛋白的分子动力学计算。提供多种类型的磷脂膜结构,同时支持自定义膜类型;
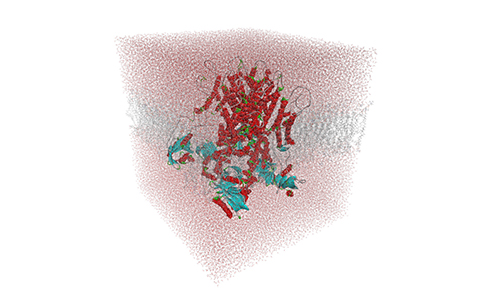
图2:在POPC显式膜内溶剂化的hERG离子通道
l 优化了分子动力学的计算步骤,每一个计算步骤都可以通过作业提交系统进行资源分配;
l QM/MM升级为64位可执行程序。
药效团(Pharmacophores):
l 新增Ensemble Pharmacophore Generation:从一组活性配体生成一组共同特征的药效团。活性配体使用2D指纹进行聚类。对于每个聚类,从聚类中心选择5个配体,然后使用共同特征药效团算法创建药效团;
l 新增Interaction Pharmacophore Generation:从受体-配体复合物的非键相互作用出发产生药效团模型;
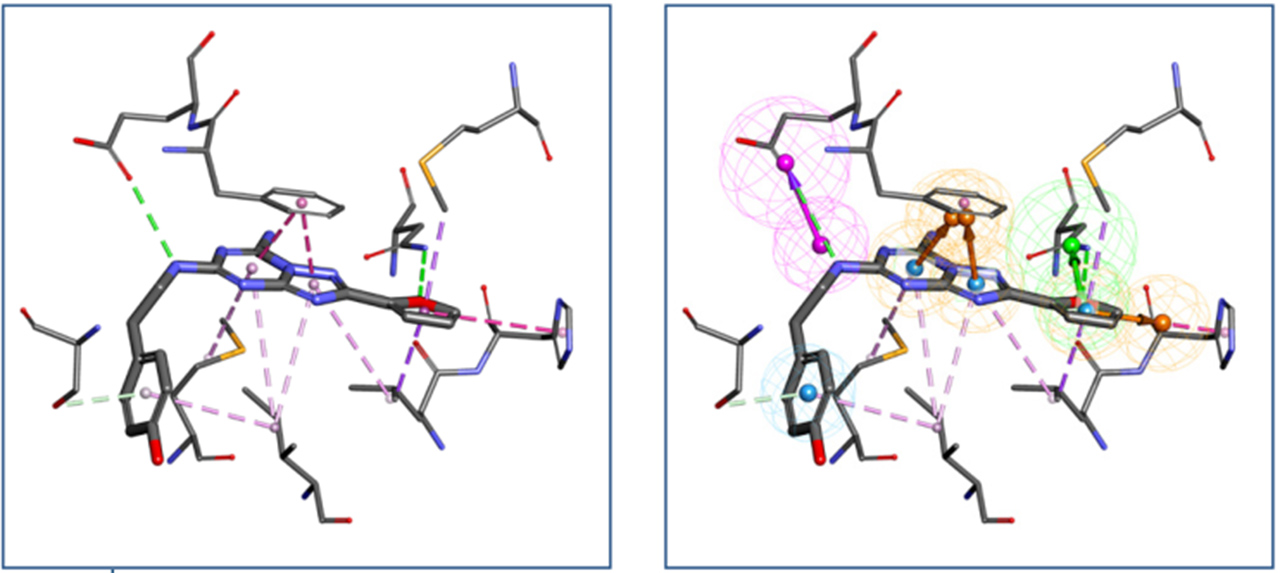
图3:依据受体-配体非键相互作用产生药效团模型
l 优化了3D QSAR Pharmacophore Generation的验证方法;
l 更新了PharmaDB数据库,容量达到253,818个药效团模型。
蛋白处理(Macromolecules):
l 优化Prepare proteins模块:可以同时处理多个蛋白分子,并添加了并行计算提高计算效率;
l 新增Analyze Protein Interface的参数Neighbor Distance Cutoff:用于确定两个氨基酸残基是否相邻。
抗体设计(Antibody):
l 新增计算抗体黏度,蛋白溶解度;
l 优化抗体人源化计算Predict Humanizing Mutations模块参数,可以同时自定义重链(H)和轻链(L)的V基因和J基因;
l 计算聚集的工具面板重命名为Predict Protein Formulation Properties。
分子对接(Receptor-Ligand Interactions):
l GOLD接口支持最新版GOLD程序,可实现共价对接功能;
l 新增核酸分子与配体分子的2D相互作用图;
l 优化了非键相互作用中配体疏水作用的定义方法。
毒性预测(TOPKAT):
l 新增解释TOPKAT计算结果的内容,帮助更好理解Toxicity Prediction(TOPKAT)和Toxicity Prediction (Extensible)计算的结果。
新增教程:
n Adding an explicit membrane to a transmembrane protein:“跨膜蛋白添加显式膜结构”教程
n Calculate relative binding free energies using Multi-Site Lambda Dynamics:“MSLD计算相对结合自由能”教程
n Generating ensemble pharmacophores from large data sets:“从一组活性配体生成一组共同特征的药效团”教程
n Generating interaction pharmacophores from a protein-ligand complex:“通过受体-配体相互作用产生药效团模型”教程
Ø 支持Pipeline Pilot 2020
Ø CHARMm版本更新至c43b2
Ø MODELER版本更新至v9.22
Ø Dmol3版本更新至v2020
Ø Blast+版本更新至v2.9
Ø 抗体模板库更新至2019年7月
Ø 客户端可以重新下载中断的作业(interrupted jobs)
Ø 客户端/服务器的操作系统
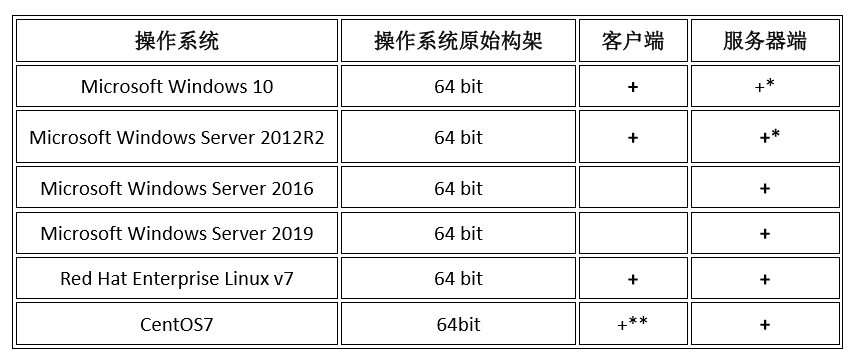
Ø 完整的Discovery Studio系统要求,请参考System Requirements for Discovery Studio 2020.
Ø 新增支持OpenSSL1.1.1c
Ø 新增支持SLURM