
抗生素通过针对入侵的生物体将自己置身于宿主-病原体的进化军备竞赛中。然而,随着病原体因自然选择进化出的耐药性,抗生素治疗效果下降。另外,抗生素的研发是一个成本高及周期长的工程,耐药性的出现促使人们对有望加快候选药物发现的计算方法产生了极大的兴趣。本文阐述了人工智能(AIDD)在小分子抗生素和抗菌肽的发现方面取得的进展。除了抗菌活性的基本预测外,还强调抗菌化合物的表征、类药性的预测、抗菌素耐药性和从头分子设计。
Ref:Commun. Biol. Published: 9 September 2021 IF=6.268
链接:https://doi.org/10.1038/s42003-021-02586-0
一、研究背景
细菌耐药性(AMR)的出现减弱了抗生素的功效,使全球发病率和死亡率水平升高。美国疾病控制和预防中心估计,美国每年有280万感染是由耐药细菌引起的,其中有3.5万人死于这种无法治疗的感染。临床试验中的抗生素也与现有的耐药机制的药物类似,进一步强调了发现全新抗生素的必要性。抗生素的研发是一个成本高及周期长的工程。2014年至2019年期间,只有14种新的抗生素成功研发并通过审批。在一项对超过21000种化合物的近186000项临床试验的调查中,研发的新药能够成功治疗传染病的可能性仅为25.2%。这种失败的风险促使企业以更高的投资回报保证进行研发,为学术界启动抗生素设计和优化的早期阶段开辟了道路。
随着人工智能和大数据的广泛应用,越来越多的科研工作者使用AIDD的方法来研发药物,有可能从根本上改变药物发现中计算模型的准确性和可靠性。可用的开源数据集(表1)、计算机工程的进步以及机器学习算法模型,极大地促进了机器学习在药物研发的应用 (AIDD),特别是抗生素发现中的应用。本文主要介绍了人工智能在小分子抗生素和抗菌肽研发中的应用。
表1与抗生素研发相关的数据库

二、化合物表征
为避免浪费大量时间用于合成非活性化合物和进行实验,研究人员通过计算来预测新设计分子的活性,在计算模型中分子的表征也是比较重要的一部分(图1)。分子的各种性质和实验可以产生大量的信息。如,为了描述简单的氨基酸残基,在线数据库中已经有400多个不同的测量方法;对于小分子药物,有计算和压缩量子力学推导出的描述符来表示拓扑性质的方法。然而,多次测量中包含的大量数据和冗余信息使得使用所有描述符变得不切实际或适得其反。因此,需要将实验数据组合成简单的描述符,以尽可能少的维度来描述尽可能多的信息。
如本文中提到可是使用AI中的图卷积网络,利用分子的几何形状和连接性将它们自然地转换成图,使用神经网络从化学结构中学习分子的特征。循环神经网络(RNN)也多用于处理分子的SMILES表示,或结合长短期记忆神经网络(LSTM)从已知药物的SMILES学习分子特征并生成新的化合物,或RNN也可与强化学习结合,根据药物的SMILES生成药物的embedding特征。此外,RNN还用于抗菌肽的表征。
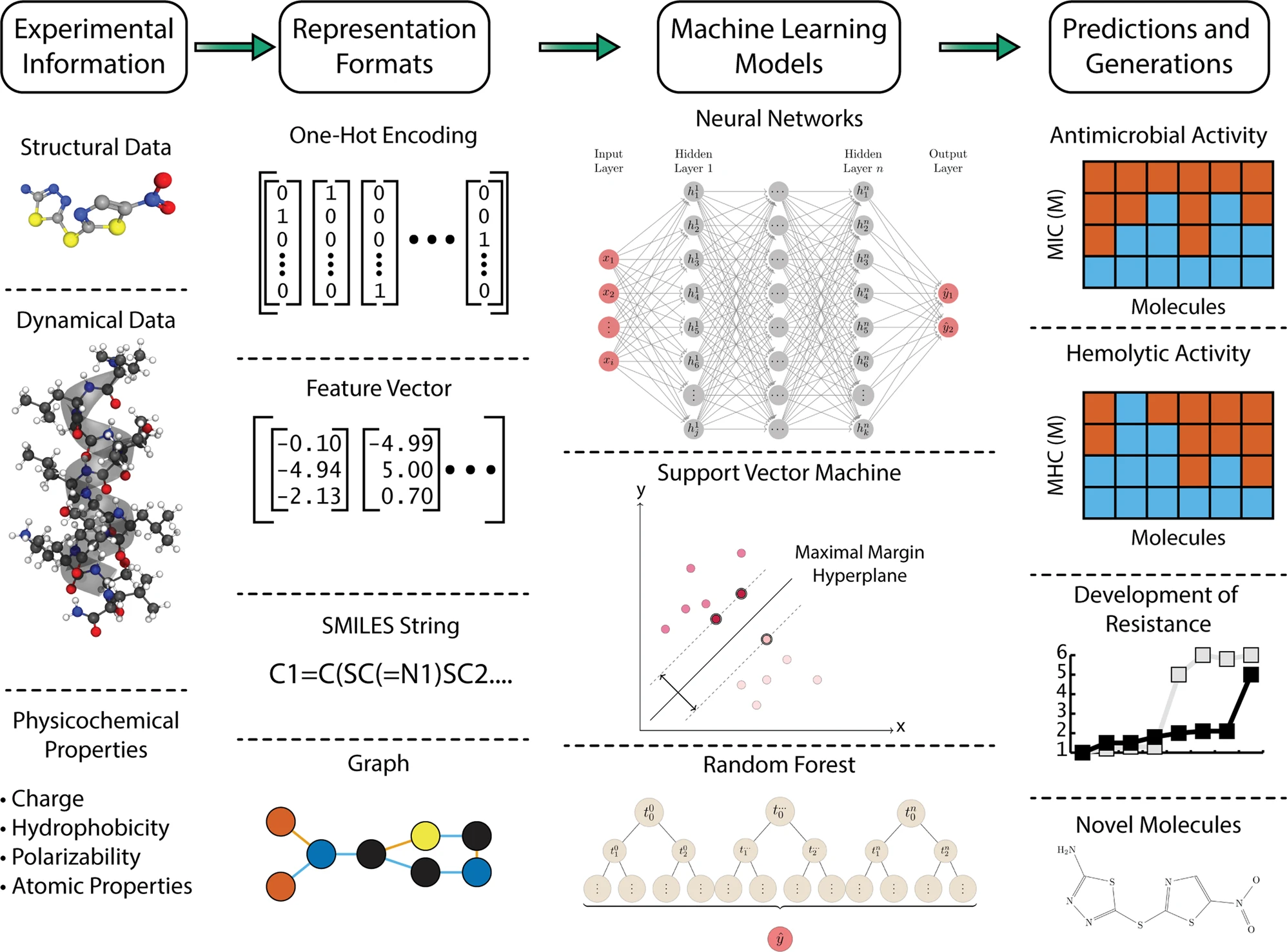
图1基于人工智能抗生素的研发方法
三、抗菌活性预测
抗菌活性的预测是AIDD整合到抗生素开发中的核心,经过多年的研究并为此提供了新的解决方案。如,研究人员使用逻辑回归对训练集中的分子片段进行分类,构造了一个基于分子片段的活性“片段库”,可以将这些分子片段拼接起来作为有活性的新抗生素;研究人员利用神经网络来表示化合物,然后评估其抗菌能力,以试图从现有的药物中选出具有抗菌作用的药物;基于支持向量机预测分子的抗菌活性;基于深度神经网络预测多肽对铜绿假单胞菌的活性;基于RNN的回归模型对抗菌肽活性的预测等方法。
四、性质预测
较差的药代动力学特性和毒性是临床试验失败的主要原因,因此迫切需要借助AIDD的方法对此进行预测。预测的性质主要包括吸收、分布、代谢、排泄和毒性(ADMET)。本文也提到有一系列工作分别利用神经网络、分类树、梯度提升分类器和共识模型预测抗菌肽和类抗菌肽药物的溶血活性。也有一系列研究利用随机森林、深度神经网络和深度泰勒分解用于预测候选药物的细胞毒性。
为考虑抗菌肽的溶解性和稳定性。已有相关工作借用神经网络、梯度增强模型、逻辑回归分类器、支持向量机和随机森林预测蛋白质溶解度。因为蛋白酶的降解也会影响抗菌肽稳定性的评估,所以可利用支持向量机、卷积神经网络、随机森林分类器和逻辑回归模型来预测水解位点,以有利于先导抗菌肽的挑选和稳定性优化。
五、耐药性预测
与大多数治疗方法不同,抗生素设计的目标是杀死具有耐药性进化能力的细菌。因此,抗生素药物设计需要考虑细菌耐药性进化。虽然当前已有基于机器学习的耐药性预测方法在临床上预测抗生素配方的耐药性,但它也可能在药物开发的实验中有用。基于耐药性基因组学的机器学习方法将越来越多地专门用于药物开发,例如利用机器学习对先导化合物的耐药性实验进行预测。目前基于机器学习的抗药性预测都是基于细菌的基因组特征,而不是药物或分子靶点特征。已有工作基于病原体基因组数据训练了能够预测细菌对抗生素的敏感性和耐药性表型的机器学习模型(AIDD)。也有工作利用SVM预测外排介导的耐药性。
虽然“黑盒”方法可能会限制AIDD在降低耐药风险的效果,但可解释性的模型可以使模型能够在机体和种群规模上找出耐药性的进化原因。将AIDD与基因-蛋白结构图谱相结合,可研究结核分枝杆菌耐药性进化的驱动因素,其假设赋予细菌耐药性的基因之间的相互作用表现为它们在SVM超平面的权重和符号的相关性。另外,也可基于蛋白质同源性的基因变异定位的可解释耐药性预测。
六、基于生成模型抗生素的发现
生成模型已经被用于化学工程和蛋白质工程,包括无机物的逆向设计和基于图的神经网络模型蛋白质折叠生成。作者着重介绍分子从头设计,其通常使用AIDD中的生成对抗网络(GANs),变分自动编码器(VAEs)相关的架构。已有工作分别利用了结合深度强化学习的生成网络,深度生成对抗自动编码器,可微分强化学习和对抗训练神经网络,结合蒙特卡罗树搜索的深度神经网络,结合随机和目标指引分子设计的自动编码器GAN用于从头药物设计(AIDD)。鉴于对序列数据的适用性,也有工作利用接受SMILES输入的RNN模型进行药物设计。如GAN已被用于生成对大肠杆菌的最低抑制浓度明显低于氨苄青霉素的抗菌肽。基于迁移学习的LSTM生成模型表明,在对较小数据集的目标特异性生物活性分子进行微调后,可以成功生成已知的靶向金黄色葡萄球菌的分子。此外,有研究者在一个基于单向LSTM的抗菌肽设计中,观察到82%的生成肽是潜在的抗菌肽,而训练数据的氨基酸分布中只有65%的随机排列被预测为抗菌素。
综之,基于AIDD的方法均可加速抗生素或抗菌肽的发现。
参考文献:
[1] Melo, M.C.R., Maasch, J.R.M.A. & de la Fuente-Nunez, C. Accelerating antibiotic discovery through artificial intelligence. Commun Biol 4, 1050 (2021).