当谈及免疫系统如何识别并对抗病原体时,T细胞和B细胞扮演着关键角色,它们通过识别特异性表位来激活免疫反应。T细胞和B细胞表位的预测对于理解免疫反应和设计疫苗至关重要。今天,我们将深入探讨B细胞结合表位和T细胞结合表位的结构特点,以及三种先进的表位预测工具——EpitopeVec、MHCflurry和DeepSeqPanII,以及如何应用三种工具实现疫苗设计。
T细胞表位结构特点
T细胞靶点预测的核心在于识别抗原中能激活CD4或CD8 T细胞的最小肽段,这一激活特性即为免疫反应性。在抗原分子中存在大量的肽片段,而T细胞靶点预测技术的目标正是筛选出那些具备激发免疫反应能力的特定肽。
作为高等脊椎动物体内最具多态性的蛋白之一,主要组织相容性复合体(MHC)拥有极其丰富的变体,IMGT/HLA数据库中记录的一类与二类MHC分子等位基因就超过了6000种。鉴于当前实验技术难以全面解析如此庞大的等位基因群体对肽的结合偏好,生物信息学预测手段成为了不可或缺的研究工具。时至今日,基于数据的分析方式已被证明是最有效的T细胞靶点预测途径。
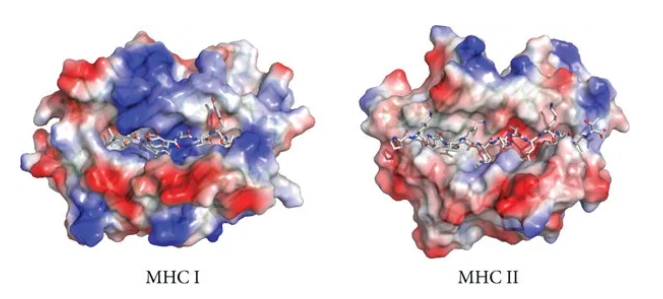
图 | MHC分子结合沟(molecule binding groove)。该图描绘了代表性MHC I和II分子的TCR所见的分子表面。MHC I分子的结合沟是闭合的,而MHC II的结合沟是打开的。结果,MHC I分子结合短肽(8-11个氨基酸),而MHC II分子结合更长的肽(9-22个氨基酸)
图片来源:
https://qinqianshan.com/biology/antibody/t-cell-b-cell-epitope-prediction/
MHC I与MHC II分子展现出近似的三维构型,但在细节上亦存显著区别。MHC I的结合槽内深藏有特定的结合口袋,仅能接纳长度介于9至11个氨基酸之间的较短肽链。大部分MHC I肽配体含有9个氨基酸,因此预测时倾向于聚焦此长度的肽。MHC II分子的肽结合槽呈现开放状态,肽段的长度可大幅变动(一般9-22个氨基酸)。
MHC-I类结合活性预测:
MHCflurry 2.0的泛等位基因模型
主要组织相容性复合体(MHC)I类分子负责在细胞表面展示内源性蛋白片段给CD8+ T细胞,是细胞介导免疫反应的重要组成部分。准确预测MHC-I类分子与其结合的多肽对于理解T细胞识别机制至关重要。MHCflurry 2.0通过整合新的MHC-I类结合模型和抗原处理模型,显著提升了预测性能。该模型利用大规模质谱识别的MHC配体数据训练抗原处理预测器,能够区分出已观察到的MHC配体和未观察到的肽段。与先前的方法如NetMHCpan 4.0和MixMHCpred 2.0相比,MHCflurry 2.0在保留数据上的表现更为出色,其综合模型优于单独组件。这种集成方法不仅改进了预测准确性,也为新抗原发现和疫苗设计提供了便捷工具。
MHC-II类结合活性预测:
DeepSeqPanII的可解释性递归神经网络
相比之下,MHC-II类分子的肽段结合预测更为复杂,因为这类分子通常识别更长且来源多样化的外源性抗原肽。DeepSeqPanII是一种针对MHC-II类肽段结合的新颖序列模型,它采用了一种带注意力机制的递归神经网络结构。与之前的方法不同,DeepSeqPanII无需对输入样本进行预处理或后处理,且通过注意力机制不仅能提供高精度的结合亲和力预测,还能揭示肽段与MHC序列间结合机制的生物学洞见。该模型通过跨等位基因的留一等位基因交叉验证和基准测试,展示了卓越的预测性能,同时,其训练数据集包括大量质谱数据和亲和力测量数据,确保了模型的广泛适用性和泛化能力。DeepSeqPanII的出现为解析HLA-II类分子的肽段结合提供了有力工具,对设计针对特定HLA-II等位基因的疫苗和免疫疗法具有重要意义。
B细胞表位的预测
B细胞表位的预测的目的在于替代完整抗原以刺激抗体生成。理论上,抗原上所有暴露于溶剂的部位均具备被抗体侦测的潜能。B细胞表位可分为两大类:线性和构象表位。线性表位由一连串未间断的氨基酸序列构成,形同肽链;而构象表位则由非连续、但均显露于溶剂中的原子群落组合而成。而且能够锁定线性表位的抗体对变性抗原有良好的辨识力,相比之下,抗原一旦变性,则会显著削弱抗体对构象表位的识别效能。

图 | 线性和构象B细胞表位。线性B细胞表位(a)由连续/连续残基组成,而构象B细胞表位(b)沿序列包含分散/不连续残基。
图片来源: https://qinqianshan.com/biology/antibody/t-cell-b-cell-epitope-prediction/
B细胞表位的预测:
EpitopeVec的深度学习方法
B细胞表位(BCEs)是位于抗原表面、能直接与B细胞受体结合的线性或构象性氨基酸序列,是激活B细胞免疫应答的关键。传统的BCEs预测方法依赖于实验数据和经验模型,但这些方法往往泛化能力有限,预测准确率仅在51%-53%之间。为克服这一限制,研究人员开发了EpitopeVec,这是一种基于深度蛋白质序列嵌入的线性BCE预测方法。EpitopeVec整合了残基属性、修改后的抗原性尺度以及基于蛋白质语言模型的表示(即蛋白质向量),并在多个大小数据集上进行了广泛验证。结果显示,EpitopeVec相较于其他前沿方法具有更高的预测性能,提高了BCE识别的准确性和可靠性,有助于快速鉴定疫苗候选物和诊断靶点。
应用案例
SARS-CoV-2疫苗设
第一步:B细胞表位预测
首先,通过B细胞表位预测工具,识别SARS-CoV-2病毒刺突蛋白(S蛋白)上的潜在线性表位。这些表位通常是病毒表面蛋白上的连续氨基酸序列,能够被B细胞受体识别,引发体液免疫反应,产生中和抗体。

图 | B细胞表位预测工作流
第二步:T细胞表位预测
接下来,使用T细胞表位预测工具,来识别S蛋白上的T细胞表位。T细胞表位的识别对于引发细胞介导的免疫反应至关重要,包括细胞毒性T细胞的激活和辅助T细胞的辅助功能。
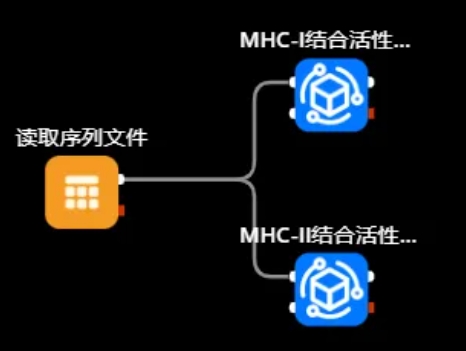
图片| MHC结合活性预测工作流
第三步:多肽-蛋白对接
在确定了潜在的B细胞和T细胞表位之后,使用多肽-蛋白对接工具AutoDock CrankPepK,来模拟这些表位肽与MHC分子的结合情况。对接模拟提供肽与MHC复合物的3D结构,评估结合的稳定性和亲和力,这一步骤对于验证表位的免疫原性和优化肽序列至关重要。

图片| 多肽-蛋白对接工作流
第四步:分子动力学模拟及结合自由能计算
经过前面几步极大缩小新抗原序列范围后,使用了分子动力学模拟手段对新抗原与MHC的结合能力进行细筛。通过模拟肽-MHC复合物在生理条件下的动态行为,可以获得关于复合物稳定性的信息,包括氢键形成、疏水作用和范德华力等。这有助于评估表位的免疫原性,即它们在真实条件下与免疫细胞受体结合的能力。
通过分子力学Poisson-Boltzmann表面面积(MM-PBSA)方法计算肽与MHC复合物的结合自由能。结合自由能计算可以量化肽与MHC分子结合的热力学稳定性,帮助筛选出最有可能引发免疫反应的表位。

图片| 多肽-蛋白复合物动力学模拟工作流
在这个全球关注健康的特殊时期,我们正站在疫苗设计与免疫学研究的前沿。你是否曾好奇过,我们的身体是如何精确地识别并击退入侵的病原体?是什么让某些疫苗如此有效,而另一些却收效甚微?
7月30日下午14点,创腾科技直播间将围绕此内容展开专题讲座,为大家更深入、更直接的讲解和演示。